El proyecto impulsado por un consorcio de más de cien científicos apunta, entre otros objetivos, a determinar si existe correlación entre características genéticas del virus y la severidad de los cuadros clínicos.
(Agencia CyTA-Leloir)-. Un consorcio de más de 100 investigadores ya secuenció cerca de 450 genomas de coronavirus provenientes de pacientes de la Patagonia, de Córdoba y del AMBA. Se van a agregar análisis genómicos de virus aislados en Chaco y Santa Fe y en próximas etapas analizarán la circulación comunitaria en todo el país.
“Nuestro objetivo es secuenciar mil genomas distribuidos en todo el país”, afirmó Mariana Viegas, investigadora responsable de genómica de virus respiratorios del Laboratorio de Virología del Hospital de Niños Dr. Ricardo Gutiérrez y coordinadora del “Consorcio interinstitucional para la secuenciación del genoma y estudios genómicos de SARS-CoV-2” que creó el Ministerio de Ciencia, Tecnología e Innovación.
La secuenciación de los genomas virales y el análisis de su evolución son importantes para conocer el patrón de circulación global y las cadenas de transmisión viral particulares, así como para monitorear cambios que podrían impactar en la biología viral, el diagnóstico molecular y la efectividad de vacunas y antivirales en los diferentes virus circulantes”, puntualizó Viegas quien también es investigadora del CONICET.
Primeros resultados
Los resultados de estos primeros análisis, realizados a partir de muestras tomadas en abril y mayo, han mostrado una “sectorización” o confinamiento en determinadas áreas geográficas de distintos linajes o grupos genéticos del virus, en virtud de la restricción del movimiento de la población, explicó Viegas.
“Por ejemplo, no encontramos un continuum del virus en Gran Buenos Aires y la Ciudad de Buenos Aires, sino que según el barrio o la localidad se podían reconocer distintos orígenes del virus”, señaló.
Sin embargo, con la flexibilización de la cuarentena y la mayor circulación de la gente, “probablemente los virus ya empezaron a mezclarse”, vaticinó. “Lo vamos a comprobar en los análisis de muestras tomadas en junio y julio”.
Una vez que se hayan obtenido todos los genomas de los coronavirus estudiados y teniendo la información clínica de los pacientes, los investigadores evaluarán si existe alguna correlación entre las características genómicas del virus y las clínicas de los pacientes que lo padecieron. “Sin embargo, hasta la fecha esto no se ha encontrado en otras partes del mundo. Hay que juntar muchos datos y casos para poder determinarla”, indicó Viegas.
El grupo de Viegas está formado por bioquímicas y biotecnólogas especializadas en genómica de virus respiratorios. “En febrero de este año, cuando vimos que la llegada de este virus podía ser posible a nuestro país, nos preparamos para tener un protocolo que pudiera secuenciar los genomas completos directamente a partir de muestras clínicas”, explicó la científica.
El consorcio interinstitucional para la secuenciación del genoma y estudios genómicos de SARS-CoV-2 cuenta con siete centros distribuidos en distintas regiones: Córdoba, Santa Fe, Neuquén, Tierra del Fuego, Buenos Aires y CABA. “La idea es generar una red de científicas y científicos que puedan responder a este problema actual pero también seguir conectados para cualquier eventual emergencia de patógenos en el futuro”.
“Coordinar este consorcio me da mucha satisfacción cuando veo que juntos vamos avanzando y generando conocimiento conjunto, que servirá para generar soberanía científica”, concluyó Viegas.

Mariana Viegas, investigadora responsable del grupo de genómica de virus respiratorios del Laboratorio de Virología del Hospital de Niños Dr. Ricardo Gutiérrez con algunas integrantes de su equipo: Laura Valinotto, Mercedes Nabaes y Mónica Natale. Coordina el “Consorcio interinstitucional para la secuenciación del genoma y estudios genómicos de SARS-CoV-2” creado por el Ministerio de Ciencia, Tecnología e Innovación.
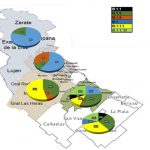
Los investigadores observaron que el linaje B.1 del coronavirus SARS-CoV-2 era el mayoritario en la Zona Norte del Gran Buenos Aires (GBA) mientras que en GBA Oeste el mayoritario era B.1.1.1 y en GBA Sur el B.1.3 y el B.1.1.33
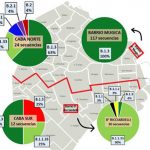
El análisis de las secuencias de los genomas de SARS-CoV-2 obtenidos en la Ciudad de Buenos Aires mostró que en la zona Norte prevaleció el linaje B1.3, único linaje detectado en el Barrio Mugica. Mientras que en la zona sur el B1.1.33.


